张 敏,博士,副教授,主要研究方向为人工智能、计算机视觉、医学图像处理.E-mail:dr.zhangmin@nwu.edu.cn.
作者简介: 
徐强强,硕士研究生,主要研究方向为机器学习、医学图像分割.E-mail:qiangqiangxu@126.com. 
任冯刚,博士研究生,主要研究方向为医工结合外科技术创新、肿瘤电磁物理消融技术.E-mail:resmak521@126.com. 
吕 毅,博士,教授,主要研究方向为肝胆胰肿瘤外科治疗.E-mail:luyi169@126.com. 
冯 筠,博士,教授,主要研究方向为医学影像处理、人工智能、模式识别、多媒体系统.E-mail:fengjun@nwu.edu.cn.
针对全自动分割算法因胰腺器官解剖变异性较高而难以实现准确定位的问题,文中提出嵌入压缩采样的编码器解码器网络.通过分阶段训练网络的方式,分割网络可级联在预训练阶段,从标签空间中感知的胰腺位置先验知识实现对分割目标的精准定位,保证分割结果与标签形状的一致性.胰腺器官分割实验表明,文中网络分割性能较优.
ZHANG Min, Ph.D., associate professor. Her research interests include artificial intelligence, computer vision and medical image processing.
AboutAuthor:
XU Qiangqiang, master student. His research interests include machine learning and medical image segmentation.
REN Fenggang, Ph.D. candidate. His research interests include medical engineering combined with surgical technology innovation and tumor electromagnetic physical ablation technology.
LÜ Yi, Ph.D., professor. His research interests include surgical treatment of hepato-biliary-pancreatic tumor.
FENG Jun, Ph.D., professor. Her research interests include medical image processing, artificial intelligence, pattern recognition and multimedia system.
Due to the high anatomical variability of pancreas, it is difficult for automated segmentation algorithms to achieve accurate localization of the target. To solve this problem, an encoder-decoder network embedded with compressive sampling is proposed. By training the network in different stages, the segmentation network can cascade the prior knowledge of pancreas location perceived from the label space in the pre-trained stage. Thus, the precise positioning of the pancreas is realized and the consistency between the segmentation result and the label is ensured. The experimental results of pancreas segmentation show that the performance of the proposed network is better.
本文责任编委 辛景民
Recommended by Associate Editor XIN Jingmin
医学图像分割技术在病理分析、临床诊断、手术动态规划等计算机辅助诊断方面具有广泛的研究意义和应用价值[1, 2, 3, 4].随着数字医疗和深度学习等领域的发展, 较大器官如心脏、肺、肾脏、肝脏等的分割技术已达到较高的精度[5, 6, 7, 8, 9, 10, 11].然而, 由于电子计算机断层扫描(Computed Tomography, CT)影像的噪声、局部体效应和组织运动的影响等因素, 胰腺器官周围存在大量紧密相连、密度相似的邻近组织, 且胰腺本身具有解剖变异性较高、占比较小等特点[4, 8, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22], 因此, 目前基于深度学习的腹部CT影像胰腺半自动分割算法和全自动分割算法的结果仍无法满足临床需求.
半自动的胰腺分割算法在训练网络时仅使用胰腺所在的最小图像块, 进行胰腺分割时需依赖医生根据先验知识手工地从CT图像中勾画包含胰腺的最小矩形框[15, 20, 22, 23].Liu等[24]提出将最小矩形区域送入U型网络(U Network, U-Net)的方法.U-Net网络包含提取上下文信息的收缩路径和与收缩路径对称的扩张路径, 能够用较短的训练时间得到较高的分割性能.Li等[20]基于残差的多尺度感受野网络, 提出多尺度跨域的信息融合策略, 有效解决过分割、欠分割及形状不一致等问题.Ning等[25]提出深度循环对抗网络(Deep Recurrent Adversarial Net- work, DRAN), 在扩大网络感受野的基础上, 建模相邻CT之间的上下文相关性, 利用对抗模块提高网络的泛化性能.
全自动分割算法能从整幅原始CT切片图像中自动分割目标器官, 摆脱对医生手工勾画的包含胰腺的最小矩形框的依赖, 辅助临床医生, 提高诊断效率.Roth等[12]提出将自底向上多层深度卷积网络(Deep Convolutional Networks, ConvNets)方法, 用于胰腺分割.Zhao等[17]提出两阶段训练的基于三维卷积神经网络(3D Convolutional Neural Network, 3D CNN)的胰腺分割网络, 先在下采样后训练一个3D U-Net网络, 得到候选区域, 再在候选区域训练另一个3D U-Net网络并分割.Man等[19]基于可形变U-Net的深度Q学习网络, 提取胰腺器官的各向异性特征, 通过交互上下文信息准确分割胰腺.总之, 全自动分割算法通常先找到目标感兴趣区(Region of Interest, ROI)后再进行细粒度的分割.相比半自动分割算法, 全自动分割算法因对目标位置和网络初始化值具有敏感性, 因此面临更大的难点和挑战[26, 27, 28, 29].
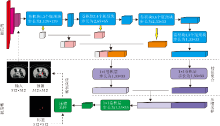
本文提出嵌入压缩采样的编码器解码器网络, 分两个阶段训练网络.第一阶段对编码器解码器网络进行预训练, 求解在压缩采样模型中定义的压缩矩阵和重建矩阵.在网络训练过程中, 通过迭代优化使压缩矩阵能提取关于胰腺位置先验的稀疏性特征表示, 网络能从标签空间中感知胰腺器官的位置先验知识.在第二阶段进行训练的分割主网络的解码器模块级联在第一个阶段求得的重建矩阵上.重建矩阵能从特征图中选择关于分割目标位置信息的稀疏性特征表示, 在胰腺的分割预测概率图重构过程中实现对胰腺器官的精准定位, 并从中恢复与原始输入图像保持相同分辨率的分割预测概率图.
针对腹部CT胰腺器官全自动分割任务, 本文提出嵌入压缩采样的编码器解码器网络.网络主要由编码器模块、解码器模块、压缩采样模块和特征聚合模块构成.
编码器模块提取关于图像的信息特征和抽象知识表示, 编码器以ResNet-50为骨干网络, 提取图像的语义特征, 添加更多的卷积层, 构成卷积块和瓶颈块, 用于增强编码器在深层提取特征的稀疏性.图像的纹理、对比度等空间局部特征包含在浅层特征图中, 深层包含图像的上下文信息和语义的表示.解码器模块通过对特征图进行上采样, 将特征空间映射到像素空间, 对特征进行重构, 恢复至与原始输入图像保持相同的分辨率, 得到胰腺的分割预测概率图.将浅层的空间局部特征与深层的语义特征进行融合[30, 31, 32, 33, 34, 35], 可减少分割目标的细粒度信息的丢失, 保留器官的形态结构, 在胰腺边缘区域保证分割结果与标签的一致性.
将分辨率大小为512×512的原始CT切片作为编码器模块的输入, 使用2层卷积核大小为3×3, 步长为2×2的卷积层提取图像特征, 经过批量归一化层(Batch Normalization, BN)和整流线性单元(Rec- tified Linear Unit, ReLU)非线性激活层对每个卷积层的输出特征图进行变换, 得到大小为128×128的高分辨率浅层特征图.卷积块中的瓶颈块交替使用的卷积核的大小为1×1或3×3, 步长为1×1或2×2, 后接BN和ReLU非线性激活层对特征进行变换, 利用最大池化层进行下采样.相比单个卷积层, 瓶颈块的使用能避免冗余卷积层的堆叠, 具有压缩特征表示的作用, 同时在网络训练过程中能有效减少参数量.
在解码器模块中, 使用大小为1×1、步长为1×1的卷积核对深层语义特征图进行变换, 实现跨通道知识交融, 对网络特征图进行通道数降维和升维的控制, 减少卷积核参数和运算复杂度.在解码器模块中嵌入压缩采样模块, 实现对胰腺位置的感知, 并将特征图恢复至与原始输入图像保持相同的分辨率, 得到胰腺的分割预测概率图.为了保留胰腺的空间形状和结构信息, 在编码器模块与解码器模块之间添加特征聚合模块, 进行跨尺度的特征整合, 提高编码器解码器网络的表达能力和分割性能.
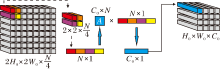
在解码器网络最后一层嵌入压缩采样模块, 如图1所示.压缩采样模块能从标签空间包含的冗余信息中感知胰腺器官的位置先验知识, 在将特征图进行恢复重构胰腺的分割预测概率图的过程中实现对分割目标的精准定位, 从而减弱网络对分割目标位置和权重初始值的敏感性, 保证分割结果与标签的形状一致性.
胰腺的位置先验知识包含在标签空间所含的冗余信息中, 采用独热编码方式对标签编码, 得到
G∈{0, 1, 2, …, C}H×W,
记为G∈{0, 1}H×W×C, 其中, H、W分别表示标签图的高和宽, C表示所有分割类别.由于胰腺分割任务只判定每个像素点属于胰腺区域或背景区域, 因此C的值为2.
编码器最后一层输出的特征O∈
N=r×r×C, r=「
在训练网络时, H/r或W/r若不是整数, 可通过双线性插值方式适当缩放原始图像和标签图像.将信号vij按照先水平后竖直的方向铺展, 构成标签预测概率图
现做如下假设:若分割目标区域在图像中占比较小, 即Gij的多数项目为0, 则重建的原始信号vij是稀疏的.通常情况下
Avij=oij.
如图2所示, 压缩矩阵A∈
即将重建矩阵B∈
arg
当原始信号vij不完全是稀疏的或观测向量oij数据存在一定偏差时, 样本空间中数据带有噪声e的干扰, 其中:
oij=Avij+e, e=[e1, e2, …, eN]T∈RN×1,
噪声e服从正态分布e~N(0, δ2)且令其满足边界条件‖ e‖ 2≤ ε , 从而保证压缩采样重建的稳定性.
为了减少图像空间信息和结构特征的丢失, 保留并融合胰腺器官的形状、轮廓和大小特征, 在编码器模块与解码器模块之间添加特征聚合模块.使用双线性插值方式, 对编码器模块中的浅层卷积块提取的特征图不断进行下采样, 将特征图的分辨率缩至与深层语义特征图保持相同, 实现跨卷积块的信息交互和特征融合.可在对任意瓶颈块中的任意卷积层提取特征进行下采样后, 与特征图O进行拼接串联融合.上述跨层级、跨通道、跨尺度的特征聚合策略, 能保留输入图像的全局结构信息, 并使之与分割目标的知识语义信息融合, 进一步提高编码器解码器网络的表达能力和性能.
如图1所示, 在第2阶段训练的分割主网络的编码器使用ResNet-50作为骨干网络, 加入更多卷积层, 增强网络的特征提取能力.以服从N(0, 0.12)高斯分布对其进行初始化, 使用第一阶段求解的重建矩阵B对使用特征聚合策略得到的特征图O进行上采样, 恢复至与原始输入图像保持相同分辨率, 得到预测概率图.第2阶段在对编码器解码器主网络进行迭代训练时, 采用交叉熵损失L(φ(X), G)优化网络参数W, 其中, G表示标签图像, 映射函数φ表示从原始输入图像X中推理得到胰腺器官的预测概率图
Φ ={φ|
优化风险函数可表示为最小化关于假设空间中的映射函数φ平均意义下的交叉熵损失L(φ(X), G), 即
由此得到嵌入压缩采样的编码器解码器网络框图, 如图2所示.
在公开NIH数据集[12]和ISBI 2015 VISCERAL挑战赛数据集[3]上进行实验.NIH数据集包含82套人体CT扫描增强数据, 每个人体的CT扫描体素的维度为512×512×L, 其中L∈[181, 466], 为沿着人体长轴方向的轴状面切片数目, 2D切片的分辨率大小为512×512.ISBI 2015 VISCERAL挑战赛数据集用于基准化多器官(肝脏、脾脏、肾脏、胰腺、胆囊、左右肾上腺等)分割, 本文仅应用其中18套有效的胰腺器官注释的人体CT扫描增强数据集, 每套CT扫描体素的维度为512×512×L, 其中L∈[194, 477].CT切片窗宽窗位范围调整为-100~240 Hu以适用于胰腺分割, 本文将含有胰腺器官的腹部CT切片作为有效数据集, 用于训练或验证.
网络训练主要分为2个级联的阶段:第1阶段求解压缩采样模型, 第2阶段训练胰腺分割主网络.为了求解压缩采样模型, 第1阶段以标准的ResNet-50为编码器骨干网络, 使用在ImageNet上的预训练权重进行初始化, 批量大小为4, 在训练集上经过2期的训练, 求解压缩采样模块中的压缩矩阵A和重建矩阵B.在第2阶段训练嵌入压缩采样模块的编码器解码器分割主网络时, 解码器除以ResNet-50骨干网络, 添加更多卷积层并令其参数初始化时服从N(0, 0.12)的高斯分布, 初始化学习效率为0.007, 动量设置为0.9, 衰减权重为0.000 1, 批量大小为4, 采用随机梯度下降(Stochastic Gradient Descent, SGD)优化器最小化交叉熵损失.在批量预处理过程中, 对原始腹部CT切片图像采用随机水平翻转的数据增强策略.分割主网络在较大的NIH数据集上训练300期, 学习率衰减呈指数下降趋势, 而在相对较小的ISBI 2015 VISCERAL 挑战赛数据集的训练集上训练1 000期, 学习率衰减采用多项式策略, 衰减权重为0.000 1.
采用四折交叉验证的方式训练和测试本文网络, 以平均准确率(mean Accuracy, mA)、平均交并比(mean Intersection over Union, mIoU)和平均Dice分数(Dice's Similarity Coefficient, DSC)作为分割性能评价指标.mA衡量分割算法处理类别不平衡的有效性, mIoU用于评估在训练过程中对分割目标进行定位的性能.令nij为将属于第i类的像素分为第j类的像素数目, nc表示总类别数目, 属于第i类像素的总数目ti=
mA=
X表示测试数据集, 假设f(xi)表示像素点xi的预测值, g(xi)表示该像素点相应的真实值, 则
DSC(f(X), g(X))=
DSC用于衡量图像预测值f(X)与真实值g(X)之间的相似性, 量化胰腺器官的分割精度.
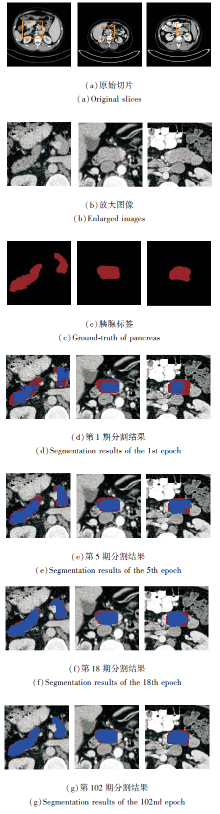
图3为本文网络随机可视化部分CT数据2D切片的胰腺分割示例.(a)中橘色矩形框是胰腺器官所在区域, (b)为胰腺所在区域的放大图像, (c)中红色区域为医生勾画的胰腺标签, (d)~(g)中蓝色区域分别表示本文网络训练到第1、5、18、102期时分割胰腺器官的结果.由(d)可见, 本文网络利用压缩采样模块从标签空间中感知胰腺的位置先验, 只需经过1期的训练, 就完成对胰腺器官的精准定位, 减弱网络对分割目标位置和权重初始值的敏感性, 解决胰腺全自动分割算法难以对较小分割目标进行定位的问题.
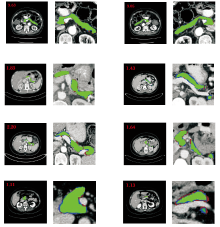
在NIH数据集上对本文网络进行训练并接近收敛时, 图4随机展示部分2D切片的分割结果, 第1列和第3列表示标签与预测值在整幅CT切片中的重叠区域(绿色区域), 左上角的红色数字表示胰腺的占比大小, 第2列和第4列为相应区域的放大图.由图可看出, 即使胰腺的位置和大小不固定、解剖变异性较高, 利用跨层级、跨通道、跨尺度的特征聚合策略, 仍能在胰腺边缘区域促使分割结果与标签趋于吻合.
本文网络在NIH数据集上的收敛趋势对比如图5所示.由图可知, 在NIH数据集上, 损失函数随迭代次数的增加迅速并趋于稳定收敛.经过前50期的训练之后, 本文网络对目标定位、对不平衡数据处理及对胰腺分割的性能达到稳定.
多期数的训练是为了更新网络参数, 使其在胰腺器官边缘区域出现的欠分割和过分割现象在可控范围内, 让近似解更逼近真实解, 进一步提高网络的鲁棒性和稳定性.
图6为本文网络在NIH数据集上第73号CT胰腺的分割结果.(a)为当网络参数训练到最优时第73号病人CT的第125、139、147、162和183张CT切片, 由图可见, 本文网络在病人CT切片上的分割结果与标签的形状具有一致性.
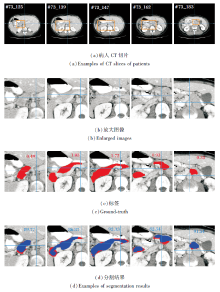 | 图6 本文网络在NIH数据集上第73号CT胰腺的分割结果Fig.6 No.73 CT pancreas segmentation results of the proposed network on NIH dataset |
图7为本文网络在NIH数据集上的胰腺3D重建结果, (b)中蓝色数字为DSC.由图可见, 本文网络能保留器官的3D基本几何结构和特征.
 | 图7 本文网络在NIH数据集上的胰腺3D重建结果Fig.7 Three-dimensional ground-truth and 3D reconstructed results of pancreas on NIH dataset |
选择如下方法进行对比:文献[3]方法、文献[5]方法、固定点模型(Fixed-Point)[6]、文献[7]方法、双阶段的三维卷积神经网络(Two-Stage 3D CNN)[8]和文献[22]方法.各方法在NIH数据集上的实验结果如表1所示.由表可见, 本文网络平均值最高, DSC最小值有较大幅度的提升, 这为网络应用于临床时的安全性提供依据, 标准差降至4.00%以下, 进一步保证网络的稳定性.
| 表1 不同方法在NIH数据集上的实验结果对比 Table 1 Experimental results comparison of different methods on NIH dataset % |
相关的胰腺分割方法在数据量相对较少的ISBI 2015 VISCERAL挑战赛数据集上的平均DSC如下:U-Net[12]为33.6%, 优势点森林(Vantage Point Forests, VPF)[4]为52.1%, 联合标签融合的多阿特拉斯方法(Multi-altas Segmentation with Joint Label Fusion, MAS-JLF)[13]为40.7%, 多数表决的多阿特拉斯方法(Multi-altas Segmentation with Majority Voting, MAS-MV)[13]为31.1%, 知识辅助卷积神经网络(Knowledge-Aided Convolutional Neural Net-work, KaCNN)[13]为58.8%, 本文网络为72.17%.由此可见本文网络的平均DSC有较大幅度提升.
本文针对腹部CT胰腺全自动分割任务, 提出嵌入压缩采样的编码器解码器网络, 将依靠数据驱动的分割网络与基于先验知识构建的压缩采样相结合, 通过两阶段级联的方式使分割网络具备器官的位置先验感知功能.本文网络能实现对分割目标的精准定位, 减弱网络对分割目标位置和权重初始值的敏感性, 保证胰腺分割结果与标签的形状一致性.在NIH、ISBI 2015 VISCERAL数据集上的实验表明, 本文网络的性能较优.今后考虑将本文网络推广应用到腹部CT多个小器官的全自动分割中.
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|
| [32] |
|
| [33] |
|
| [34] |
|
| [35] |
|