卜起荣,博士,副教授,主要研究方向为计算机视觉、医学图像处理、机器学习.E-mail:boqirong@nwu.edu.cn.
作者简介: 
张墺琦,硕士研究生,主要研究方向为医学图像处理、机器学习、深度学习.E-mail:zhangaoqi2018@yeah.net. 
亢宇鑫,博士研究生,主要方向研究为医学图像处理、机器学习、深度学习.E-mail:yuxinkang@stumail.nwu.edu.cn. 
武卓越,硕士研究生,主要研究方向为计算机视觉、机器学习.E-mail:buz2559@dingtalk.com. 
崔 磊,博士,讲师,主要研究方向为计算机视觉、医学图像处理、深度学习.E-mail:leicui@nwu.edu.cn.
针对肝脏组织病理图像分割中存在的正常组织和异常组织过渡区域较难分割和空洞较多的问题,设计基于多尺度特征和注意力机制的肝脏组织病理图像语义分割网络.在编码器中提取融合多尺度特征,改善正常组织和异常组织过渡区域的分割效果.同时利用注意力机制对空间维度和通道维度进行相关性建模,获得每个像素类内响应和通道间的依赖关系,缓解肝脏组织病理图像空洞较多对网络学习带来的影响.实验表明文中网络可较快速准确分割肝脏组织病理图像损伤区域.
BU Qirong, Ph.D., associate professor. His research interests in-clude computer vision, medical image proce-ssing and machine learning.
AboutAuthor:
ZHANG Aoqi, master student. Her re-search interests include medical image proce-ssing, machine learning and deep learning.
KANG Yuxin, Ph.D. candidate. His re-search interests include medical image proce-ssing, machine learning and deep learning.
WU Zhuoyue, master student. His research interests include computer vision and machine learning.
CUI Lei, Ph.D., lecturer. His research in-terests include computer vision, medical image processing and deep learning.
To address the problem of difficult segmentation and many voids in the transition regions of normal and abnormal tissues in liver histopathology images segmentation, a semantic segmentation network of pathological images of liver tissue based on multi-scale feature and attention mechanism is proposed. The fused multi-scale features are extracted in the encoder to improve the segmentation of the transition regions between normal and abnormal tissues. The attention mechanism is employed to model the correlation between spatial dimension and channel dimension to obtain the response of each pixel within each class as well as the dependency between channels, and the impact of many voids in liver histopathology images on the network learning is alleviated. Experiments demonstrate that the proposed network can segment the damaged regions of liver histopathology images more quickly and accurately.
本文责任编委 桑农
Recommended by Associate Editor SANG Nong
肝脏损伤是常见疾病, 多由酒精性肝中毒、服用药物不当等导致.为了治疗和研究人类肝脏损伤, 需要显微镜下的组织病理分析.由于病理切片场景较大、背景复杂、组织区域众多、人工观察费时, 并且病理切片分析难度较高, 需要长期经验积累, 不同经验的病理学家分析结果可能有差别.随着人工智能技术的快速发展, 利用扫描设备将病理切片数字化, 借助人工智能算法对数字病理切片进行自动分析成为研究热点[1, 2, 3, 4, 5, 6], 主要集中在利用深度学习模型实现病理图像的分割、分类、检测任务.对病理图像的良好分割有助于病理学家对异常组织区域或细胞进行定量分析, 也可提高后续检测模型结果的准确率.
病理图像分割任务包括语义分割、实例分割和全景分割, 其中病理图像的语义分割是对图像中的每个像素分类, 实现正常区域和病变区域的分割.Tokunaga等[7]使用卷积神经网络(Convolutional Neu-ral Networks, CNN)作为二进制像素分类器, 用于标记每个像素, 实现肺腺癌病理图像中不同癌症亚型区域的分割.病理图像的实例分割在语义分割的基础上实现对每个目标对象进行区分, 但它不区分背景, Qu等[8]提出全分辨率CNN, 实现对细胞核和腺体区域的实例分割.病理图像的全景分割是语义分割和实例分割的结合, 在像素级分类的同时区分每个目标, Zhang等[9]提出端到端的病理图像全景分割网络, 实现对病理图像中癌细胞的全景分割.
肝脏病理图像分割作为病理图像分割中的一个方向, 也取得一定进展.Okada等[10]运用统计图谱和统计模型对病理图像中的肝血窦进行自动分割.Cruz-gomez等[11]利用传统的图像处理方法, 提出基于颜色直方图和多尺度形态学滤波等融合的肝脏细胞分割算法, 在肝脏组织切片H& E染色(Hema-toxylin-Eosin Staining)图像的细胞核和细胞膜分割中, 取得85%的细胞分割正确率.Rojas-moraleda等[12]提出基于计算拓扑框架的生物医学图像中细胞核的鲁棒检测和分割方法, 并在肝脏组织病理图像上进行肝脏细胞检测, 检测准确率达到86.2%, 分割Dice系数为0.5.Roy等[13]通过高曲率点检测和椭圆拟合质量评估方法对肝脏病理图像中的脂肪变性区域进行分割和分离, 分割脂肪变性区域的准确率为86%.
上述肝脏组织病理图像的分割方法主要基于数学形态学, 对数据预处理要求较高, 若预处理工作不彻底, 则需要进行一系列的基于点的开(闭)运算, 运算速度明显下降.Xu等[14]研究大肠癌肝细胞转移, 利用多尺度特征结合深度卷积神经网络(Deep CNN, DCNN), 对肝组织病理图像块进行癌变区域的分割, 得到96%的定量分析准确率, 但方法对数据集要求较高, 需要不同放大倍数下的病理图像和对应标注, 网络参数量较大, 需要大量训练样本.
目前对肝脏组织病理图像分割存在如下难点:1)肝脏正常组织区域与异常组织区域的交界处特征差异较小, 难以准确区分; 2)已有的数据集样本较少, 在提供给模型有限可学习知识的情况下, 如何确保训练模型的准确率和鲁棒性是一个问题; 3)相比其它组织病理图像, 肝脏组织病理图像因其组织结构的特殊性, 图像中空洞较多, 对分割模型的学习造成一定干扰.针对问题1)和3), 研究者们常采用通过融合在不同放大倍数下的图像提取的特征增加上下文语义信息的方式改善过渡区域的分割效果.Xu等[14]采取此方法提高肝脏组织病理图像分割精度, 然而目前数据集较小, 不易获取, 并且这种方法需要不同放大倍数下的图像和对应标注, 增加获取数据的难度.对于问题2)一般采取翻转、色彩抖动、生成对抗网络(Generative Adversarial Network, GAN)等数据增强的方式扩充数据集[14, 15, 16], 这种方式从医学角度讲可解释性较弱.
为了解决上述肝脏组织病理图像分割难点, 本文提出基于多尺度特征提取融合和注意力机制的肝脏组织病理图像语义分割网络, 帮助病理专家提高阅片效率, 为病理专家进行下一步定量定性分析提供支持.本文网络利用空洞卷积[17]提取不同感受野的特征并融合, 增加上下文语义信息, 在一定程度上解决肝脏组织病理图像分割中的边界难分问题.受Non-local模块[18]和分割中使用的注意力机制[19]的启发, 本文网络分别对位置和通道维度进行注意力建模, 获得空间维度和通道维度的特征相关性, 进一步提高模型对正常组织区域、异常组织区域和背景的区分能力, 缓解肝脏组织病理图像空洞多对模型学习带来的影响.为了解决数据样本较少带来的过拟合问题, 采用U型网络(U-Net)的基础架构作为主干网络[20], 使网络参数量较少, 在样本数量较少时不易过拟合, 提高模型的鲁棒性.在此基础上利用跳跃连接, 结合用于定位的深层特征和用于精确分割的浅层特征, 适用于解决肝脏组织病理图像边界模糊的问题.
为了处理肝脏组织病理图像分割难点, 本文分别从两方面出发:一方面利用空洞卷积扩大网络的感受野, 融合不同空洞率提取的特征图[17, 21], 获得更多的细节信息, 改进难分边界的分割效果; 另一方面利用空间和通道上的注意力机制建模改进编码器, 获得特征的全局依赖信息, 缓解肝脏组织病理图像中空洞对模型学习造成的干扰.
本文网络结构图如图1所示, 由4个下采样模块、1个特征注意力模块、1个多尺度特征提取融合模块和4个上采样模块组成.每个下采样模块由1个双卷积层和最大池化层组成, 双卷积层由2个3×3卷积层、批归一化(Batch Normalization, BN)[22]层、组归一化(Group Normalization, GN)[23]层、ReLU激活函数组成.本文网络在双卷积操作中采用批归一化和组归一化结合的方式, 解决BN操作数据太少时计算的均值和方差不准确的问题.输入的图像尺寸为512×512, 由于显存的限制, BN中批数据量不能太大.为了消除批数据量太小的影响, 选取BN与GN结合的方式, 加快收敛, 提高性能.不同于BN, GN是对通道进行划分, 计算均值和方差, 文献[23]已证实不会受到批数据量的影响.
本文网络的注意力模块由位置注意力模块和通道注意力模块组成.多尺度特征提取融合模块采用类似空间金字塔池化结构[17, 21].上采样模块由双线性插值和特征拼接组成, 采用双线性插值将输入特征上采样至与跳跃连接特征大小相同的尺度, 拼接两个特征, 将双卷积操作后得到的特征输入下一个上采样模块中, 重复上述操作, 直至恢复原图大小.
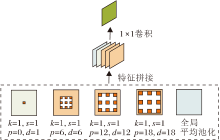
为了在现有放大倍数的肝脏组织病理数据基础上获得更多的上下文信息, 改善肝脏组织病理图像中正常组织和异常组织过渡区域的分割效果, 根据Chen等[21]提出的ASPP(Atrous Spatial Pyramid Poo-ling)模块思想, 利用不同膨胀率的空洞卷积并行地提取多尺度特征, 融合提取的多尺度特征图.本文网络使用的多尺度特征提取和融合模块具体结构如图2所示, 图中k为卷积核尺寸, s为步长, p为填充范围, d为膨胀率, 分别使用膨胀率为6、12、18的空洞卷积[21, 24, 25]提取特征.为了防止空洞率过大可能导致卷积退化, 将其与全局平均池化后的特征进行融合.此模块的使用增大感受野, 获得更多的上下文信息, 有助于改善模型对边界细节的分割能力, 在一定程度上解决肝脏病理图像中异常组织和正常组织边界难分问题.
本文将不同膨胀率提取的不同尺度的特征进行拼接, 然后采用1×1的卷积, 转化为固定大小的特征图, 由于融合不同尺度的特征, 因此提高网络对肝脏病理图像中小区域病变的分割能力, 同时也减少特征信息的损失, 有助于后续解码器上采样恢复细节信息.
当前基于深度学习的病理图像语义分割网络通常采用多尺度特征融合[7, 14]或U-Net的结构[20]融合低层和高层的语义特征的方式, 未综合考虑特征图各个位置或通道之间的联系和相关性.因此本文网络分别对特征图的位置维度和通道维度进行相关性建模, 首先使用Non-local[18]结构实现对特征空间位置的相关性建模, 再使用类似Non-local的结构对通道间相关性进行建模, 并将两个模块的输出结果进行相加融合, 获得特征间的全局依赖关系, 提高网络对正常组织区域、异常组织区域和背景的区分能力, 改善正常组织和异常组织过渡区域的分割效果, 同时在一定程度上缓解空洞多对网络学习带来的不利影响.
肝脏组织病理图像中不同组织区域内的细胞形状、染色情况等存在差异, 但组织间也存在一定的拓扑结构相似性, 为了利用不同区域特征之间的关联, 相互增强各自特征的表达, 本文网络利用类似Non-local中的建模方式对位置进行建模, 如图3所示.首先计算任意两点特征之间关联强度矩阵, 即原始特征A经过卷积降维获得特征B(维度为(H×W)×C')和特征C(维度为C'×(H×W)), 进行矩阵乘积, 获得任意两点特征之间的关联强度矩阵, 维度为(H×W)×(H×W).经过softmax归一化操作, 获得每个位置对其它位置的注意力图S, 越相似的两点特征之间响应值越大.将位置注意力图中响应值作为加权对特征D进行加权融合, 通过像素间的关联性达到选择性增强或抑制特征的作用.
对于特征图S∈RN×N:
Sij=
其中, C为特征通道数, H、W为特征图的高和宽, N=H×W.Sij为第i个位置对第j个位置的影响, 也就是第i个位置和第j个位置之间的关联程度/相关性, 越大越相似.
位置注意力模块输出特征图E∈RC×H×W:
Ej=α
其中:α为尺度系数, 初始化为0, 通过逐渐学习分配更新权重; E为所有位置特征相似度和原始位置的加权和, 具有全局上下文信息.
不同通道间的特征图可看作是对特定类别的响应, 不同响应间具有一定的依赖关系.为了显式地建模这种依赖关系, 受Non-local模块启发, 本文网络对通道维度进行类似的注意力建模, 如图4所示.
通过建模通道之间的关联, 增强通道下特定语义响应能力.具体过程与位置注意力建模相似, 不同的是在获得特征注意力图X∈RC×C时, 将任意两个通道特征进行维度变换和矩阵乘积, 得到任意两个通道的关联强度.同样经过softmax操作获得通道间的注意力图.通过通道之间的注意力图加权进行融合, 使各个通道之间产生全局关联, 获得更强语义响应的特征.具体计算过程如下:
xij=
其中, C为特征通道数, H、W为特征图的高和宽, xij为第i个通道对第j个通道的影响, β 为尺度系数, 初始化为0.通过迭代学习更新权重, 最终得到的输出E是每个通道特征和原始特征的加权和.本文通过类内特征响应、类间特征抑制的方式, 强化网络对边界的敏感程度.
肝脏组织病理图像分割流程如图5所示, 对肝脏组织病理切片进行数字化, 得到全场图像, 由于全场图像大小基本都大于10 000×10 000, 计算机无法直接对其进行处理.本文采用滑动窗口策略, 将全场图像分为大小为512×512的图像块, 输入到本文网络中, 得到正常组织区域和异常组织区域后, 根据专家需求计算这个图像块上异常区域占组织的比值, 实现定量分析.
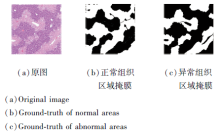
数据集源自国内某医院病理科采集的30幅H& E染色肝脏组织病理全场病理切片图像, 采用滑动窗口策略从中截取197组大小为512×512的样本图像块用于标注.如图6所示, 白色区域为专家标注区域, 每幅对应有正常区域的掩膜和坏死区域的掩膜, 实验使用159组作为训练集, 38组作为测试集.
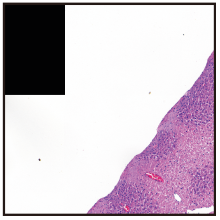
本文网络输入的图像块由肝脏组织病理全场图像通过滑动窗口策略生成.对于在窗口滑动生成图像块过程中出现的裁剪至边界的情况, 采用填充的方法对图像进行外补0操作, 得到512×512的图像块.初步实验发现, 填补0后的黑色区域(如图7所示)会对模型学习带来干扰, 因此在实验前对数据集上的黑色区域图像进行预处理, 将黑色区域和背景统一转化为255后进行实验.
本文实验使用的电脑CPU为Intel Core i7-8700K, 内存为16 GB, GPU为Nvidia GeForce GTX 1080Ti, 使用PyTorch 1.4.0版本开源库训练和测试网络模型, 操作系统为windows10.
采用随机梯度下降算法(Stochastic Gradient Descent, SGD)对网络参数进行优化, 网络参数的初始化采用Xavier[26], 初始学习率为0.1, 权重衰减设定为0.000 5, 使用多项式衰减策略进行训练过程中的学习率衰减, 即当前学习率等于初始学习率乘以(
本文使用交叉熵作为损失函数:
L=-
其中, pi表示预测结果中第i个像素的概率, gi表示金标准的第i个像素的真实类别, N表示像素数量.
具体评价指标如下.
1)Dice系数.描述真实标注的掩模和预测图之间的相似性:
d=
2)像素分类准确率(Accuracy of Pixels Classi-fication, AP).所有分类正确的像素数占像素总数的比例:
AP=
3)频权交并比(Frequency Weight Intersection over Union, FWIoU).根据每类出现的频率设置权重, 权重乘以每类的交并比(Intersection over Union, IoU)并进行求和:
FWIoU=
为了证实模块的有效性, 进行模块消融实验, 定义如下模块.U-Net+CA表示单独使用注意力模块中的通道注意力(Channel Attention, CA)部分改进U-Net编码器.U-Net+PA表示单独使用注意力模块中的位置注意力(Position Attention, PA)部分改进U-Net编码器.U-Net+DA表示使用完整的注意力模块(Dual Attention Module, DA)改进U-Net编码器.U-Net+MFF表示使用多尺度特征提取融合模块(Multi-scale Feature Extraction and Fusion, MFF)改进U-Net编码器.
各模块的消融实验结果如表1所示.由表可知, 无论是单独使用注意力模块还是使用多尺度特征融合模块改进编码器, Dice系数、PA、FWIoU值都得到一定提升.
| 表1 各模块的消融实验结果 Table1 Ablation experiment results of different modules |
图8为各模块消融实验的可视化结果, 在图中, 白色区域为正常组织区域, 灰色区域为异常组织区域, 黑色区域为背景.由图可更明显地看出不同模块的改进作用, 相比U-Net, 使用注意力模块改进后边界分割效果对异常区域的分割更准确, 并在一定程度上缓解分割后不准确的空洞现象.这是由于注意力建模融合特征图上每个像素对不同类的响应和通道之间的依赖性, 对像素的分类更准确.本文单独对注意模块中的通道注意力和位置注意力进行实验, 相比完整的注意力模块, 在细节上有一定差距, 这是由于完整的注意力模块对通道注意力和位置注意力得到的注意力图进行相加融合, 使特征在一定程度上得到互补.
相比U-Net, 使用多尺度特征提取融合模块改进编码器在边界细节方面更完善, 这是因为融合多尺度特征可获得更大的感受野和更多的上下文信息, 更有利于解码器上采样恢复边界细节.
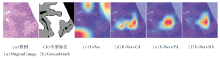
同时, 为了研究注意力模块的作用, 本文使用梯度类激活映射方法(Visual Explanations from Deep Networks via Gradient-Based Localization, Grad-CAM)[27]将特征进行可视化, 结果如图9和图10所示, 其中, 颜色越红的区域表示此处激活值越大, 即模型越关注.由图可知, 在对异常区域进行分割预测时, U-Net关注的区域有部分异常区域、少部分正常区域和背景.将U-Net分别与CA、PA结合后, 模型更关注异常区域.而在U-Net同时引入通道注意力模块和位置注意力模块(即U-Net+DA)后, 在对异常区域的预测过程中, 模型不仅更关注异常区域且更完整, 可见本文的注意力模块在对异常组织区域进行分割时, 可提高模型对于异常区域的关注度.
由图10可见, 在正常组织区域进行分割预测时, U-Net关注的区域较分散, 有正常区域、异常区域及背景, 而在使用本文注意力模块后模型更关注正常区域.
综上所述, 本文的注意力模块比不使用或单独使用通道/位置注意力模块更有助于网络关注需要分割的区域, 提高对肝脏组织病理图像的分割精度.
 | 图9 网络预测异常区域时Grad-CAM可视化特征结果Fig.9 Grad-CAM visualization feature results with abnormal areas predicted network |
在肝脏组织病理图像数据集上将DASU-Net与常用于医学图像的语义分割网络对比.具体对比网络如下:基于注意力的U-Net医学图像(胰腺为例)分割网络(Attention U-Net: Learning Where to Look for the Pancreas, AttU-Net)[28], 基于U-Net的递归残积卷积神经网络(Recurrent Residual CNN Based on U-Net, R2U-Net)[29], 用于医学图像分割的嵌套式U-Net架构(UNet++: A Nested U-Net Architec-ture for Medical Image Segmentation, Nested-UNet)[30], 基于可分离卷积的编码-解码结构(En-coder-Decoder with Atrous Separable Convolution for Semantic Image Segmentation, Deeplabv3 plus)[31].实验结果如表2所示, 由表可看出, 相比U-Net, 本文网络在坏死组织区域Dice系数提高4.5%.
| 表2 各网络实验结果对比 Table 2 Experimental result comparison of different networks |
各网络的可视化结果如图11所示, 白色区域表示正常组织区域, 灰色区域表示异常组织区域, 黑色区域表示背景.由图可见, U-Net对于坏死区域和正常区域的分割效果都不佳.相比U-Net, AttU-Net分割性能得到明显提升, 这得益于AttU-Net在编码器和解码器上都使用注意力机制, 不过还是存在坏死区域分割不准确的问题.R2U-Net和NestedUNet对于坏死区域和正常区域也存在不能准确区分二者边界的问题, NestedUNet对于正常区域的分割效果优于R2U-Net.由于使用基于深度残差学习的图像识别网络(Deep Residual Learning for Image Recog-nition, ResNet)[32]和基于深度可分卷积的深度学习架构(Deep Learning with Depthwise Separable Convolutions, Xception)[33]作为基础网络, Deeplabv3 plus网络层数较多, 参数量较大, 不利于使用样本量较少的数据集训练, 但其引入空间金字塔池化模块进行多尺度特征的提取和融合, 对边界细节恢复还是有一定帮助, 相比UNet、AttU-Net、R2U-Net、NestedUNet和专家标注, Deeplabv3 plus边界更光滑.本文网络无论在正常区域还是在坏死的异常区域, 分割效果都得到明显提升, 尤其是图中框出区域.这得益于编码器分别对空间维度和通道维度基于注意力机制建模得到特征间的相关性, 及多尺度的特征提取融合获得更多的上下文信息, 同时网络采用跳跃连接结构, 最大限度地融合高级特征和低级特征, 解码器对称地进行上采样, 也最大限度地恢复特征信息.
本文提出基于多尺度特征和注意力机制的肝脏组织病理图像语义分割网络, 在语义分割常用的编码解码结构下, 使用注意力模块和多尺度特征提取融合模块改进编码器.首先提取多尺度特征并进行融合, 获得更多的上下文信息, 改善对小区域病变和边界的分割效果.然后利用注意力机制分别对每个特征图进行相关性建模, 得到每个像素对不同类的响应, 同时在通道间进行建模, 得到通道之间的依赖
关系.最后将两个维度建模得到的特征进行融合, 得到既有类间依赖关系又有通道相关性的特征, 改善对肝脏坏死区域分割中空洞和过渡区域不易分割的问题.对比实验表明, 本文网络实现较快速、准确的肝组织病理图像坏死区域分割, 解决常用的医学影像分割网络对于肝组织病理图像中边界分割不准确、空洞较多的问题, 在评价指标上明显优于其它常用于病理图像分割的网络.此外, 本文网络由于参数量较少且浅层特征与深层特征结合较充分的原因, 适用于样本量较少的肝坏死数据集, 与目前常用于组织病理图像分割网络相比, 在同样不进行数据增强的情况下开展实验, 获得较优性能, 相比通过数据增强后增加数据量训练获得的模型, 本文网络的医学可靠性更高.下一步将在其它组织病理图像数据集上进行实验, 测试本文网络在病理图像上的泛化性.
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|
| [32] |
|
| [33] |
|