董守斌,博士,教授,主要研究方向为高性能计算、生物信息学和生物计算、自然语言处理等.E-mail: sbdong@scut.edu.cn.
作者简介: 
郑潇柔,博士,助理研究员,主要研究方向为高光谱遥感图像、半监督学习、跨域迁移学习.E-mail: zhengxrsc@scut.edu.cn. 
卢汝诺,硕士研究生,主要研究方向为计算机视觉、遥感图像.E-mail:202220143179@mail.scut.edu.cn. 
刘 凯,硕士研究生,主要研究方向为深度学习、计算机视觉、图像识别.E-mail:202321043978@mail.scut.edu.cn.
不同成像模式设备采集的医学图像存在不同程度的分布差异,无监督域自适应方法为了将源域训练的模型泛化到无标注的目标域,通常是将差异分布最小化,使用源域和目标域的共有特征进行结果预测,但会忽略目标域的私有特征.为了解决该问题,文中提出基于目标域增强表示的医学图像无监督跨域分割方法(Enhanced Target Domain Representation Based Unsupervised Cross-Domain Medical Image Segmentation, TreUCMIS).首先,通过共有特征学习获取源域和目标域的共有特征,通过图像重构训练目标域特征编码器,提取目标域完整特征.然后,通过目标域的无监督自学习方式,加强深层特征和浅层特征的共有性.最后,对齐使用共有特征和完整特征得到的预测结果,利用目标域的完整特征分割目标,提高模型在目标域的泛化性.在两个具有CT和MRI双向域自适应任务的医学图像分割数据集(腹部、心脏)上的实验表明TreUCMIS的有效性与优越性.
DONG Shoubin, Ph.D., professor. Her research interests include high performance computing, bioinformatics and biocomputing, and natural language processing.
About Author:
ZHENG Xiaorou, Ph.D., assistant professor. Her research interests include hyperspectral remote sensing imagery, semi-supervised learning and cross-domain transfer learning.
LU Runuo, Master student. Her research interests include computer vision and remote sensing imagery.
LIU Kai, Master student. His research interests include deep learning, computer vision and image recognition.
Medical images produced by different imaging modality devices exhibit varying degrees of distribution differences. Unsupervised domain adaptation methods typically aim to generalize models trained in the source domain to the unlabeled target domain by minimizing these distribution differences and using shared features between the source and target domains for result prediction. However, they often neglect the private features of the target domain. To address this issue, a method for enhanced target domain representation based unsupervised cross-domain medical image segmentation(TreUCMIS) is proposed in this paper. First, TreUCMIS acquires common features through shared feature learning, and a target domain feature encoder is trained through image reconstruction to capture the complete features of the target domain. Second, unsupervised self-training of the target domain strengthens the shared characteristics of deep and shallow features. Finally, the predicted results obtained from the shared and complete features are aligned, enabling the model to utilize the complete features of the target domain for segmentation and thus improving the generalization in the target domain. Experiments on two medical image segmentation datasets involving bidirectional domain adaptation tasks with CT and MRI(abdominal and cardiac datasets) demonstrate the effectiveness and superiority of TreUCMIS.
近年来, 深度学习技术在医学图像分割领域表现出优越性能.然而, 深度学习技术需要使用大量的有标注数据进行训练, 在一个域(即源域)上训练且表现良好的模型, 其性能在另一个未知域(即目标域)上可能会表现不佳或性能不稳定, 出现域偏移(Domain Shift, DS)问题.DS问题在医疗领域尤为突出, 由于不同医疗机构之间数据采集设备的差异、扫描参数的不同以及患者群体的多样性, 医学图像数据在不同域之间呈现出复杂的域分布差异, 导致模型在源域训练后难以泛化到目标域[1, 2].
解决域偏移的一种直接方法是在大量源域数据上预训练深度学习模型, 再使用少量带标签的目标域数据进行微调[3].然而, 医学图像标记过程需要专业的知识和经验, 获取带有像素级标注数据的成本极高.相比之下, 无监督域自适应(Unsupervised Domain Adaptation, UDA)方法可利用有标注的源域数据和未标记的目标域数据训练深度学习模型, 提高模型在目标域的泛化性.
现有的无监督域自适应方法主要分为基于图像对齐的无监督域自适应方法、基于特征空间对齐的无监督域自适应方法、基于输出空间对齐的无监督域自适应方法.基于图像对齐的无监督域自适应方法[4, 5, 6]利用对抗生成网络进行图像到图像的风格转换, 将源域图像风格转换为目标域图像风格, 用于对齐源域图像和目标域图像的外观.有研究者利用CycleGAN(Cycle Generative Adversarial Network)[6], 将源域图像风格转换为目标域图像风格, 并结合其它方法, 进一步提高模型在目标域的性能[7, 8].基于特征空间对齐的无监督域自适应方法[9, 10]一般通过对抗学习迫使网络最小化目标域和源域的特征分布差异, 学习源域和目标域的共有特征.例如:ACDA(Attention-Based Cross-Layer Domain Alignment)[9]和PnP-AdaNet(Plug-and-Play Adversarial Domain Adapta-tion Network)[10]在多个尺度上对齐源域和目标域的特征空间, 解决域偏移问题.基于输出空间对齐的无监督域自适应方法[11, 12]一般通过对抗学习, 对齐源域和目标域的预测结果, 在输出空间对齐预测结果可避免高维特征的复杂性, 提升模型对特征的解码能力.
基于图像对齐的无监督域自适应方法通常需要大量源域和目标域数据学习风格转换过程, 效率较低, 并且进行风格转换后的图像对结构信息的保存能力较弱, 不利于语义分割任务.基于特征空间对齐的无监督域自适应方法可直接在特征级别对齐源域和目标域, 避免图像转换而丢失结构信息的问题.然而目前该方法大多专注于学习域不变特征, 可能忽略目标域的私有特征在语义分割中的潜在价值, 例如:忽略不同器官之间、器官与背景之间的边界颜色差异等信息, 使分割目标的边界识别不准确或部分目标难以识别.在基于特征空间对齐的无监督域自适应方法中, 会使用源域数据进行有监督学习, 对齐后的特征不可避免地倾向源域, 导致模型在目标域性能下降.基于输出空间对齐的无监督域自适应方法的对齐力度较弱, 单独使用时难以有效将模型泛化到目标域, 通常需要结合其它对齐方法一起使用.
本文提出基于目标域增强表示的医学图像无监督跨域分割方法(Enhanced Target Domain Represen-tation Based Unsupervised Cross-Domain Medical Image Segmentation, TreUCMIS).通过特征对齐的方法提取共有特征.通过一致性正则化的方法, 增强共有特征.通过自学习和一致性正则化, 使模型学会利用目标域图像的完整特征进行分割.为了避免图像级别对齐导致图像结构信息的丢失, 构建共有特征学习模块(Shared Feature Learning Module, SFLM), 学习源域与目标域的共有特征, 通过共有特征将模型泛化到目标域.为了缓解基于共有特征学习的UDA往往会导致模型更偏向具有强标注标签源域的偏向性问题, 构建共有特征增强模块(Shared Feature En-hancement Module, SFEM), 增强目标域的深层共有特征和浅层共有特征的无监督自学习, 缓解特征偏向性问题.针对当目标域与源域之间差异过大时, 忽略目标域的私有特征会严重影响模型在目标域的泛化性问题, 构建目标域泛化模块(Target Domain Ge-neralization Module, TDGM), 增强目标域的完整特征和共有特征的自学习, 使模型学会利用目标域图像的完整特征进行分割.在腹部器官数据集和心脏结构数据集上进行的MRI和CT双向交叉模态的无监督域自适应实验表明, TreUCMIS可提升未标记目标域图像的分割性能.
医学图像语义分割是指对医学图像中的不同结构和组织进行像素级别的分类, 以实现对图像的精细解析和定量分析.近年来, 深度学习技术被广泛应用于医学领域[13, 14], 深度学习技术可帮助医生提供更准确、高效的诊断和治疗方案.许多研究表明, 深度学习方法可显著提高生物组织分割的准确性[15], 如针对脑部组织的分割方法[16]、针对乳腺的分割方法[17, 18].也有学者针对腹部器官分割[19]和心脏结构分割[20]进行深入研究.尽管这些方法表现出良好的性能, 但通常需要使用带有专家标注的大型数据集, 因此方法昂贵耗时.
由于扫描仪的不同或跨模态变化, 不同医学图像之间往往存在域偏移问题.无监督域自适应方法可将学到的知识从有标记的源域泛化到无标记的目标域, 成为解决医学图像中域偏移问题的有效方法.现有的无监督域自适应方法主要分为基于图像对齐的无监督域自适应方法、基于特征空间对齐的无监督域自适应方法、基于输出空间对齐的无监督域自适应方法三类.
基于图像对齐的无监督域自适应方法旨在通过图像转换, 将一个域的图像转换为另一个域的图像, 从而减少不同域图像的外观差距.CycleGAN[6]可在没有成对示例图像的情况下, 转换源域图像风格和目标域图像风格, 并能保持图像原有的语义信息.CyCADA(Cycle-Consistent Adversarial Domain Adaptation)[4]使用CycleGAN进行源域图像和目标域图像的风格转换, 同时在高级语义空间上进行特征对齐.近年来, 基于图像对齐的无监督域自适应方法在医学图像UDA任务上得到广泛应用.Chen等[21]提出SIFA(Synergistic Image and Feature Adaption), 将CT图像与MRI图像进行风格转换, 降低CT图像和MRI图像之间的域分布差距.Zhang等[22]提出TD-GAN(Task Driven Generative Adversarial Network), 实现对未见过的真实X射线图像的同步风格迁移.Zhang等[23]在使用条件生成网络生成目标图像的同时引入形状一致性损失, 约束生成图像的形状畸变.Du等[7]提出CUDA(Constraint-Based Unsupervised Domain Adaptation Network), 使用CycleGAN进行图像风格转换, 并使用转换后的图像进行跨域自监督学习.Liu等[8]提出FSUDA, 使用基于频域和空间域转移的方法, 从源域生成类目标域图像, 并使用类目标域图像和目标域图像训练模型, 同时使用多教师蒸馏框架, 进一步提升性能.基于图像对齐的无监督域自适应方法在医学图像上取得一定成果, 但需要大量的数据训练图像转换过程, 并且在域差距较大时, 容易出现模式崩溃和训练不稳定的问题, 导致效果大幅降低.
基于特征空间对齐的无监督域自适应方法侧重于提取域不变特征, 利用对抗学习的思想, 学习域不变特征[24, 25].在医学领域, Dou等[10]提出PnP-Ada-Net, 在多层次特征上对齐, 并在医学CT图像和MRI图像上进行实验, 验证方法的有效性.Hu等[26]提出DoCR(Domain Specific Convolution and High Fre-quency Reconstruction), 构建域特定卷积模块, 提高分割模型的域不变特征提取能力.Chen等[27]提出SIFA(Synergistic Image and Feature Alignment), 在多个方面利用对抗性学习和深度监督机制, 同时跨域变换图像的外观, 增强提取特征的域不变性, 在CT图像和MRI图像之间进行无监督域自适应.Yang等[28]提出FDA(Fourier Domain Adaptation), 通过傅里叶变换与逆变换缩小源域和目标域的差距.Zhao等[29]提出LEUDA(Label-Efficient Unsupervised Domain Adaptation), 使用CycleGAN进行图像风格转换, 并使用自集成对抗学习模块进行特征对齐.尽管基于特征空间对齐的无监督域自适应方法在医学领域取得一定效果, 但容易导致对齐的特征偏向源域特征, 忽略目标域的私有特征对分割结果的影响.
基于输出空间对齐的无监督域自适应方法在源域和目标域的预测结果上进行对抗性自适应.Tsai等[11]构建多层次的对抗网络, 在不同的特征层得到预测结果, 并在多个预测结果上进行对抗性域自适应.Zeng等[5]提出ICMSC(Intra- and Cross-Modality Semantic Consistency), 首先通过CycleGAN转换不同域的图像风格, 并约束经过循环转换后的图像按照与之前完全相同的方式进行分割.Feng等[12]提出SE_ASA, 将目标域图像添加不同的噪声, 得到多组预测结果, 并在图像的输出空间进行对比, 筛选可靠像素和不可靠像素, 进行选择性熵约束.
给定源域数据集Ds=
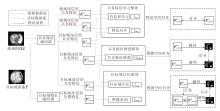
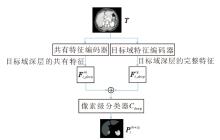
本文提出基于目标域增强表示的医学图像无监督跨域分割方法(TreUCMIS), 整体结构如图1所示.
TreUCMIS由共有特征编码器Esh、目标域特征编码器Etg、共有特征学习模块(SFLM)、共有特征增强模块(SFEM)和目标域泛化模块(TDGM)组成.特征编码器Esh和Etg均采用ResNet50结构, 但不共享权重.Esh用于提取目标域和源域的共有特征, Etg用于提取目标域图像的完整特征.Esh和Etg会同时提取图像的浅层特征和深层特征, 将ResNet50第3个stage输出的特征作为浅层特征, 特征通道数为1 024, 将第4个stage输出的特征作为深层特征, 特征通道数为2 048.通过SFLM, 方法可学习源域和目标域的共有特征, 并学会使用共有特征进行语义分割, 对目标域图像具有良好的分割能力.SFEM对比目标域共有特征中多层次特征的预测结果, 进一步增大共有特征空间, 强化解码器利用共有特征进行分割的能力.然而, 当目标域与源域存在显著差异时, 模型仅利用共有特征进行分割, 容易导致分割效果不佳, 因此本文引入TDGM, 通过图像重构训练目标域特征编码器, 提取目标域完整特征, 并将使用不同特征得到的预测结果对齐, 使模型学会利用目标域的完整特征进行分割, 提高其在目标域的泛化性.模型可用端到端的方式进行训练, 整体优化目标如下:
ltotal=lSFLM+lSFEM+lTDGM,
其中lSFLM、lSFEM、lTDGM分别表示SFLM、SFEM、TDGM损失.
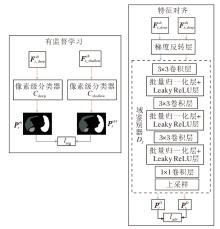
共有特征学习模块(SFLM)的目的是提取源域和目标域的共有特征, 并使模型学习利用该共有特征进行语义分割.SFLM详细结构如图2所示.在特征对齐部分, 将源域图像和目标域图像的特征输入域鉴别器中, 通过对抗学习, 混淆源域图像和目标域图像的域鉴别结果, 达到特征对齐的目的[35].然后利用对齐后的源域图像特征进行有监督学习, 使模型学会利用共有特征进行分割.
SFLM构建特征级别的域鉴别器, 实现源域和目标域的共有特征的提取.域鉴别器通过判断特征是来自源域还是目标域, 并加入梯度反转层[35], 实现类似CycleGAN的对抗损失.梯度反转层的工作原理是在前向传播时作为一个恒等操作, 即直接传递输入特征.但在反向传播时, 会将传入的梯度乘以一个负系数(如-1), 在反向传播过程中对域分类器的梯度进行反转.这样做的效果是:特征提取网络在训练过程中会尽可能地生成域鉴别器难以区分的特征, 从而使源域和目标域的特征分布更接近.域鉴别器由多个卷积层堆叠, 最后将卷积层的输出进行上采样操作, 得到逐像素的域鉴别器分类结果.具体来说, 给定源域图像
$ \begin{array}{l} \boldsymbol{P}_{s}^{D}=D_{f}\left(E^{s h}\left(\boldsymbol{x}_{i}^{s}\right)\right) \in \mathbf{R}^{H \times W}, \\ \boldsymbol{P}_{t}^{D}=D_{f}\left(E^{s h}\left(\boldsymbol{x}_{i}^{t}\right)\right) \in \mathbf{R}^{H \times W}, \end{array}$
其中Df(· )表示域鉴别器.
对于来自源域的特征, 将其域鉴别的真实标签设为1H× W.对于来自目标域的特征, 将其真实标签设为0H× W.使用交叉熵损失函数计算域鉴别器损失:
ladv=
其中, di∈ {0H× W, 1H× W}, 表示样本i的真实域标签, 0H× W表示形状为H× W的全0矩阵, 1H× W表示形状为H× W的全1矩阵.注意, 在进行特征对齐时, 即计算ladv时, 如图2所示, 使用Esh提取的多层特征中的深层特征
提取源域和目标域的共有特征后, 使用源域的有标注数据及共有特征训练模型, 使模型学会利用共有特征进行语义分割, 能较好地分割目标域图像.具体地, TreUCMIS使用共有特征编码器Esh与分割头Cshallow和Cdeep对源域图像进行有监督学习.有监督学习损失函数如下:
$ \begin{aligned} l_{\text {seg }}= & \sum_{i=1}^{N_{s}} \alpha \operatorname{Ce}\left(\boldsymbol{y}_{i}^{s}, C_{\text {shallow }}\left(E^{\text {sh }}\left(\boldsymbol{x}_{i}^{s}\right)\right)\right)+ \\ & \beta C e\left(\boldsymbol{y}_{i}^{s}, C_{\text {deep }}\left(E^{\text {sh }}\left(\boldsymbol{x}_{i}^{s}\right)\right)\right), \end{aligned}$
其中, Ce(· )表示多元交叉熵损失函数, Cshallow(· )和Cdeep(· )表示不同的语义分割头, 即像素级分类器, α 、 β 表示不同语义分割头对应的损失权重, 设置α =0.4, β =0.6.
如图2所示, 将Esh提取的多层特征中的浅层特征
lSFLM=γ ladv+lseg,
其中, γ 表示域鉴别损失的损失权重, 取值为0.6.
在SFLM中对齐源域图像和目标域图像的深层特征, 然而由于域偏移问题, 源域和目标域共有的语义信息可能会分布在不同的特征层上, 仅对深层特征进行对齐容易导致浅层特征的共有语义信息不匹配.模型在训练过程中, 会利用源域图像进行有监督训练, 共有特征会不可避免地向源域倾斜, 导致模型的解码器对共有特征的解码能力变弱.
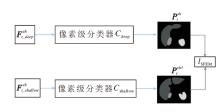
为了解决上述问题, TreUCMIS构建共有特征增强模块(SFEM).研究表明[9], 在源域和目标域的多个特征级别上进行特征对齐, 可增大各级别特征的共有语义信息.与文献[9]不同, 为了增强解码器对共有特征的解码能力, SFEM不是在特征级别上直接对齐多个层次的特征, 而是将目标域的浅层特征和深层特征分别输入不同的解码器中, 得到两组预测结果, 并在输出空间对齐这两组预测结果.
在SFLM中, 已对深层特征进行特征层面的对齐.在SFEM中, 利用不同层次的特征通过解码器得到的预测结果在输出空间进行对齐, 可约束浅层特征包含更多的共有化语义信息, 同时可加强解码器对浅层共有特征、深层共有特征的解码能力, 缓解共有特征偏向源域的问题, 从而对共有特征进行增强.
SFEM详细结构如图3所示, 通过共有特征编码器提取目标域图像的浅层特征和深层特征, 并输入不同的分割头中, 得到两组预测结果, 再在输出空间对齐这两组预测结果, 并计算一致性损失, 这样可增大目标域浅层特征、深层特征的共有空间, 同时强化模型使用共有特征预测分割掩码的能力.
具体地, 如图3所示, 将共有特征编码器Esh(
lSFEM=
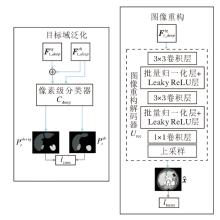
SFLM和SFEM能学到源域图像和目标域图像的共有特征, 使模型利用共有特征较好地分割目标域图像.然而, 当源域和目标域差异较大时, 仅依赖共有特征进行分割可能会忽略目标域中的关键私有特征, 导致性能下降.为了解决该问题, TreUCMIS构建目标域泛化模块(TDGM), 使模型学到利用目标域图像的完整特征进行目标分割的能力.
TDGM的详细结构如图4所示.目标域图像编码器通过图像重构任务被迫保留足够的信息重建输入图像, 这促使目标域图像编码器学到如何从输入图像中提取完整特征, 如图像纹理、颜色等特征, 并将这些特征压缩到编码空间中.TDGM首先使用图像重构技术, 使目标域特征编码器Etg能学到目标域图像的完整特征.通过卷积层与上采样层实现图像重构, 重构后的图像:
$\hat{\boldsymbol{x}}_{i}^{t}=U_{\mathrm{rec}}\left(E^{t g}\left(\boldsymbol{x}_{i}^{t}\right)\right), $
其中, Etg(· )表示目标域特征编码器, Urec(· )表示图像重构的解码器.则图像重构损失:
$l_{\mathrm{recon}}=\frac{1}{N_{t}} \sum_{i=1}^{N_{t}}\left(\boldsymbol{x}_{i}^{t}, \hat{\boldsymbol{x}}_{i}^{t}\right)^{2} .$
使用图像重构技术后, Etg学到如何提取目标域图像的完整特征.经过共有特征提取和共有特征增强后, 解码器学到如何利用共有特征对目标域图像进行分割结果预测.对于同幅目标域图像, 利用共有特征编码器和目标域特征编码器得到的特征, 解码后的预测结果应该是一致的, 利用对比学习, 在预测空间对不同特征的预测结果进行一致性正则化, 可提升解码器对目标域特征编码器提取特征的解码能力, 加强模型对目标域图像完整特征的利用, 从而使模型进一步泛化到目标域.为了使模型更好地利用源域数据和目标域数据的知识, 将由目标域的共有特征编码器Esh提取的共有特征与目标域特征编码器Etg提取的特征进行融合, 使用该融合特征与共有特征在输出空间进行对比学习.
TreUCMIS将目标域图像共有特征及目标域图像完整特征融合后, 再输入语义分割头, 得到预测结果的原因如下.在SFLM中使用大量源域数据学习源域图像和目标域图像的共有特征, 这些源域数据增强共有特征编码器Esh提取特征的能力, 目标域特征编码器Etg使用目标域数据进行图像重构训练, 能学到部分被共有特征编码器Esh忽略的目标域的私有特征, 因此共有特征编码器Esh和目标域特征编码器Etg提取的目标域图像的特征是互补的, 利用这种融合特征进行分割可提高模型在目标域的性能.采用特征相加的方式作为特征融合的方式, 融合后的特征为:
将目标域图像的共有特征、融合特征分别输入分割头Cdeep中, 得到目标域图像基于共有特征的预测结果
和目标域图像基于融合特征的预测结果:
由此得到TDGM一致性损失:
lcons=
在TDGM中, 如图4所示, 使用特征编码器得到多尺度特征的深层特征
$l_{\mathrm{TDGM}}=\delta l_{\text {recon }}+\epsilon l_{\text {cons }}, $
其中, δ 表示lrecon的损失权重, $\epsilon$表示lcons的损失权重, 设置δ =0.2, $\epsilon$=0.5.
本文选择如下两个数据集进行实验.
1)腹部器官数据集.使用与文献[27]相同的腹部多器官数据集, 包含来自ISBI2019 CHAOS(Com- bined(CT-MR) Healthy Abdominal Organ Segmenta-tion) 挑战赛[19]的20例MRI数据和来自文献[36]的30例CT数据, 两个数据集均为公开数据集, 都包含肝脏、右肾、左肾和脾脏四个器官的像素级标签, 本文目的是使用深度学习模型分割上述四种腹部器官.
2)心脏结构数据集.使用MM-WHS(Multi-mo-dality Whole Heart Segmentation)2017挑战赛数据集[37], 包含20例MRI数据与20例CT数据, 其中CT与MRI均为未配对数据.CT与MRI均包含升主动脉(Ascending Aorta, AA)、左心房血腔(Left Atrium Blood Cavity, LAC)、左心室血腔(Left Ventricle Blood Cavity, LVC)和左心室心肌(Myocardium of the Left Ventricle, MYO) 四种心脏结构的像素级标签, 本文目的是使用深度学习模型分割上述四种心脏结构.
对于腹部器官数据集和心脏结构数据集, 随机划分80%数据作为训练数据, 20%数据作为测试数据.对于腹部器官数据, 采用与SIFA相同的预处理方法, 截取腹部区域, 并去掉器官周围无关的背景区域, 采用z-score转换, 所有的图像被归一化到平均值为0、标准差为1, 每幅图像被重采样至256× 256, 最后以扫描的轴向视图对图像进行2D切片.对于心脏结构数据, 在冠状面上裁剪以心脏为中心的区域, 并截断顶部2%的强度直方图以减轻剪影, 采用z-score转换, 所有的图像被归一化到平均值为0、标准差为1, 每幅图像被重采样至256× 256, 最后以冠状视图对图像进行2D切片.
对于腹部器官数据集和心脏结构数据集, 将2D切片扩展为三通道图像再输入网络中, 这是因为模型的编码器使用ImageNet预训练的ResNet50权重.在训练阶段, 使用随机旋转、随机裁剪、颜色变换数据增强, 颜色变换表示随机改变图像的对比度、饱和度和亮度.在源域上使用随机旋转、随机裁剪、颜色变换数据增强, 在目标域上仅使用随机旋转、随机裁剪数据增强.
与FSUDA[8]、SIFA[27]相同, 本文使用Dice相似系数(Dice)和平均对称表面距离(Average Sym- metric Surface Distance, ASD)评估方法分割性能.Dice衡量预测掩码和真实掩码之间的体素精度.ASD衡量预测掩码和真实掩码体素表面之间的平均距离.Dice值越高, ASD值越低, 表明分割效果越优.Dice计算公式如下:
Dice=
其中, TP表示真阳性的数量, FP表示假阳性的数量, FN表示假阴性的数量.
ASD的计算公式如下:
$\begin{aligned} A S D= & \frac{1}{2|Y|} \sum_{y \in Y} \min _{p \in P} d(y, p)+ \\ & \frac{1}{2|P|} \sum_{p \in P} \min _{y \in Y} d(p, y), \end{aligned}$
其中, Y表示真实分割标注的边界点集合, P表示预测分割结果的边界点集合, y表示真实分割标注边界上的点, p表示预测分割结果边界上的点, d(· , · )表示欧氏距离,
TreUCMIS主要由二维卷积网络组成.共有特征编码器Esh和目标域特征编码器Etg具有相同的架构, 均采用ResNet50, 不共享权重.本文的深层特征Fdeep采用ResNet50最后一个stage的输出, 通道数为2 048.浅层特征Fshallow采用ResNet50第3个stage的输出, 通道数为1 024.
鉴别器Df由4个卷积层组成, 前3个卷积层的卷积核为3× 3, 最后一个卷积层的卷积核为1× 1, 步幅为1, 通道数为{256, 128, 32, 2}.除了最后一个卷积层, 每个卷积层后都连接一个批量归一化层和Leaky ReLU层, 将最后一层卷积层的输出上采样至和输入图像一样的大小.
图像重构解码器Urec由3个卷积层组成, 前2个卷积层的卷积核为3× 3, 最后一个卷积层的卷积核为1× 1, 步幅为1, 通道数为{256, 64, 3}, 除了最后一个卷积层, 每个卷积层后连接一个批量归一化层和Leaky ReLU层, 将最后一层卷积层的输出上采样至和输入图像一样的形状.
分割头Cshallow采用FCN(Fully Convolutional Networks)的分割头, Cdeep采用DeepLabv3的ASPP(Atrous Spatial Pyramid Pooling)分割头, 图2~图5中Cdeep共享权重, Cshallow共享权重.
基于pytorch1.7.1实现TreUCMIS, 在RTX2080Ti上进行实验.在预训练阶段, 设置迭代次数为20, 批尺寸大小为8, 采用AdamW(Adaptive Moment Esti-mation with Decoupled Weight Decay)优化器, 优化器中β =0.9, 0.999, 权重衰减参数为0.01, 学习率为0.000 06.在正式训练阶段, 设置迭代次数为20, 批尺寸大小为6, 采用SGD(Stochastic Gradient Descent)优化器, 优化器的动量参数为0.9, 权重衰减参数为1e-4, 其中图像重构解码器Urec、Cshallow和Cdeep的学习率为0.005, 其余模块的学习率为0.000 5.
选择如下对比方法:CyCADA[4]、ICMSC[5]、Cycle-GAN[6]、CUDA[7]、FSUDA[8]、文献[11]方法、SIFA[27]、FDA[28]、LE-UDA[29].其中, SIFA、FSU-DA、LE-UDA、CUDA使用的数据集与本文相同, 本文直接引用原论文的实验结果, 其它对比方法的实验结果来自文献[8]和文献[27].
本文还对比不使用无监督域自适应的结果, 即W/o adaptation, 作为分割性能的下界, 该结果是使用预训练阶段在源域数据上训练完成的网络, 直接对目标域数据的测试集进行测试得到的.本文使用目标域数据有监督训练的Supervised作为分割性能的上界, 该结果是使用预训练阶段的网络架构, 在目标域数据带标注的训练集上训练模型, 并在目标域数据的测试集上进行测试得到的.
本文分别在腹部器官CT→ MRI(即源域数据为腹部器官CT图像, 目标域数据为腹部器官MRI图像)、腹部器官MRI→ CT、心脏结构CT→ MRI、心脏结构MRI→ CT分割任务上进行实验.
各方法在腹部器官CT→ MRI分割任务上的性能对比如表1所示, 在腹部器官MRI→ CT分割任务上的性能对比如表2所示, 表中黑体数字表示最优值.由表可见, 在不进行域自适应的情况下, 方法对MRI分割的平均Dice值仅为74.4%, 对CT分割的平均Dice值仅为77.0%.相比有监督训练结果, MRI相差18.3%, CT相差14.9%.这表明在腹部器官数据的CT和MRI间有较严重的域偏移.使用TreUCMIS后, 相比W/o adaptation:对MRI分割的平均Dice值提升16.0%, 平均ASD值降低0.9; 对CT分割的平均Dice值提升8.3%, 平均ASD值降低2.8.这说明TreUCMIS可有效缓解域偏移问题.
| 表1 各方法在腹部器官CT→ MRI分割任务上的性能对比 Table 1 Performance comparison of different methods in abdominal organ CT→ MRI segmentation tasks |
| 表2 各方法在腹部器官MRI→ CT分割任务上的性能对比 Table 2 Performance comparison of different methods in abdominal organ MRI→ CT segmentation tasks |
在无监督域自适应CT→ MRI分割任务中, FSUDA表现最优, 平均Dice值为89.2%, 平均ASD值为1.5, 而TreUCMIS的平均Dice值为90.4%, 平均ASD值为1.3, 相比FSUDA, 平均Dice值提高1.2%, 平均ASD值降低0.2.LE-UDA在ASD评分上表现最优.
在无监督域自适应MRI→ CT分割任务中, LE-UDA表现最优, 平均Dice值为84.4%, 平均ASD值为1.6, 而TreUCMIS的平均Dice值为85.3%, 平均ASD值为1.3, 相比 LE-UDA, 平均Dice值提高0.9%, 平均 ASD值降低0.3.
值得注意的是, 在腹部器官分割任务中, 相比其它方法, TreUCMIS的优势并不显著, 可能是因为腹部器官数据集的域偏移问题并不特别严重.TreUCMIS是在域不变性模型基础上的改进, 当域偏移不是特别严重时, 性能提升不明显.
图6可视化不同方法对腹部器官的分割结果, 分别使用黄色、红色、棕色、蓝色表示肝、右肾、左肾、脾, 图像左上角红色方框为细节放大图.由图可见, 使用不同的域适应方法后, 分割结果得到很大改善, TreUCMIS取得更精细的分割结果.例如:观察图6第2幅图像和第3幅图像发现, 只有TreUCMIS分割出左肾.综上所述, TreUCMIS在腹部器官分割任务中表现出较优性能.
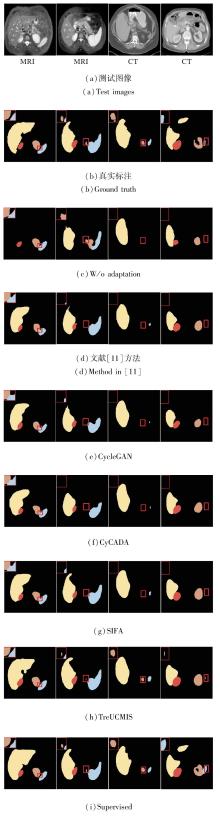 | 图6 各方法在腹部图像上分割结果的可视化Fig.6 Visualization of segmentation results of different methods on abdominal images |
各方法在心脏结构CT→ MRI分割任务上的性能对比如表3所示, 在心脏结构MRI→ CT分割任务上的性能对比如表4所示, 表中黑体数字表示最优值.由表可见, 在不进行域自适应的情况下, 方法对MRI分割的平均Dice值仅为49.1%, 相比有监督训练结果, MRI相差35.0%.在不进行域自适应的情况下, 方法对CT分割的平均Dice值仅为65.2%, 相比有监督训练结果, CT相差28.9%.上述结果说明, 心脏结构的CT图像和MRI图像间存在较严重的域偏移问题, 比腹部器官数据的域偏移问题更严重.使用TreUCMIS后, 对MRI分割的平均Dice值由49.1%升至79.5%, 提升30.4%, 平均ASD值降低7.8.对CT分割的平均Dice值由65.2%升至90.7%, 提升25.5%, 平均ASD值降低7.1.
| 表3 各方法在心脏结构CT→ MRI分割任务上的性能对比 Table 3 Performance comparison of different methods in cardiac structure CT→ MRI segmentation tasks |
| 表4 各方法在心脏器官MRI→ CT分割任务上的性能对比 Table 4 Performance comparison of different methods in cardiac structure MRI→ CT segmentation tasks |
在无监督域自适应CT→ MRI分割任务中, LE-UDA表现最优, 平均Dice值为69.8%, 平均ASD值为3.5, 而TreUCMIS的平均Dice值为79.5%, 平均ASD值为3.2, 相比LE-UDA, 平均Dice值提高9.7%, 平均ASD值降低0.3.
在无监督域自适应MRI→ CT的分割任务中, FSUDA表现最优, 平均Dice值为85.3%, 平均ASD值为2.6, 而TreUCMIS的平均Dice值为90.7%, 平均ASD值为1.4, 相比FSUDA, 平均Dice值提高5.4%, 平均ASD值降低1.2.
由表1~表4可发现, 心脏结构数据集的域偏移问题比腹部器官数据集更严重, TreUCMIS在心脏数据上对域偏移问题的改善更显著, 这表明在域偏移更严重的情况下, TreUCMIS的改善效果更明显.
图7可视化不同方法对心脏结构的分割结果, 分别使用黄色、红色、棕色、蓝色表示MYO、LAC、LVC、AA的心脏结构, 图像左上角红色方框为细节放大图.
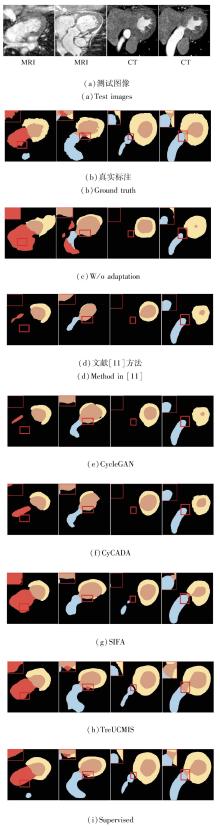 | 图7 各方法在心脏图像上分割结果的可视化Fig.7 Visualization of segmentation results of different methods on cardiac images |
由图7可见, 使用不同的域适应方法后, 分割结果得到很大改善, TreUCMIS取得更精细的分割结果, 对器官位置及器官边界的分割更准确.例如:在图7第2幅图像中, TreUCMIS成功分割左心房血腔(红色部分).综上所述, TreUCMIS在心脏结构分割任务中表现较优性能.
本文在腹部器官MRI→ CT分割任务和心脏结构CT→ MRI分割任务上进行消融实验, 验证TreUCMIS中SFLM、SFEM、TDGM的有效性, 具体结果如表5所示, 表中黑体数字表示最优值.
| 表5 各模块的消融实验结果 Table 5 Ablation experiment results of different modules |
由表5可见, 当不使用任何域自适应方法时, 对腹部器官分割的平均Dice值仅有77.0%.使用SFLM进行共有特征提取后, 平均Dice值比W/o adaptation提升7.1%, 达到84.1%.当不使用任何域自适应方法时, 对心脏结构分割的平均Dice值仅有49.1%.使用SFLM进行共有特征提取后, 平均Dice值比W/o adaptation提升28%, 达到77.1%.这充分说明SFLM在提取MRI与CT共有特征上的有效性.
在SFLM的基础上, 使用SFEM后, 腹部器官分割的平均Dice值提升0.2%, 达到84.3%.心脏结构分割的平均Dice值提升0.3%, 达到77.4%.这说明SFEM增强多层次特征共有空间的有效性.
在SFLM的基础上, 使用TDGM后, 腹部器官分割的平均Dice值提升0.5%, 达到84.6%, 心脏结构分割的平均Dice值提升1.0%, 达到78.1%.这说明TDGM学习目标域完整特征进行分割的有效性.
结合SFLM、SFEM、TDGM, 腹部器官分割的平均Dice值达到85.3%, 比W/o adaptation提高8.3%, 心脏结构分割的平均Dice值达到79.5%, 比W/o adaptation提高30.4%.结合SFLM、SFEM、TDGM后性能优于使用单个模块, 这是因为结合SFLM和SFEM增强共有特征空间后, 预测结果更准确, TDGM使用增强后的共有特征, 在输出空间进行一致性正则化, 对模型的训练更稳定.
为了进一步验证提取的目标域的融合特征对分割性能提升的有效性, 对比在使用SFLM、SFEM与TDGM训练完模型后, 使用共有特征和融合特征进行分割的效果.在腹部器官分割中使用目标域的融合特征进行分割比使用目标域的共有特征进行分割的平均Dice值提高1.5%, 在心脏结构分割中使用目标域的融合特征进行分割比使用目标域的共有特征进行分割的平均Dice值提高1.8%.
实验表明, 使用目标域的共有特征和目标域的完整特征融合后的特征进行分割, 效果更优.在心脏结构分割中, 域偏移问题比腹部器官更严重, 且应用各个模块的提升比腹部器官更大, 说明在域偏移更严重的数据集上, TreUCMIS的优势更明显.
相比FSUDA、LE-UDA, 本文使用颜色变换数据增强.下面对其进行消融实验, 在心脏结构数据集和腹部器官数据集上分析W/o adaptation情况下使用颜色变换数据增强及UDA情况下使用颜色变换数据增强对性能的影响, 结果如表6和表7所示.表中使用ctrans(Color Transformation)表示颜色变换数据增强, 黑体数字表示最优值.
| 表6 腹部器官分割任务数据增强消融实验结果 Table 6 Results of ablation experiments on data augmentation for abdominal organ segmentation tasks |
| 表7 心脏结构分割任务数据增强消融实验结果 Table 7 Results of ablation experiments on data augmentation for cardiac structure segmentation tasks |
由表6和表7可见, 在腹部器官和心脏结构CT→ MRI分割任务中, 不使用UDA, 使用颜色变换数据增强, 可显著提升分割的准确性.但在MRI→ CT分割任务中, 不使用UDA, 使用颜色变换数据增强对分割性能的提升较小.这说明在不进行域自适应的情况下, 特别是在CT→ MRI分割任务中, 颜色变换数据增强可提升方法对目标域的分割性能, 可能的原因是颜色变换数据增强使方法学到源域和目标域的部分共有特征.
在不使用颜色变换数据增强的情况下使用TreUCMIS, 比单独使用颜色变换数据增强的平均Dice值提高5%以上, 而结合颜色变换数据增强和TreUCMIS, 分割效果得到进一步提升.综上所述, 颜色变换数据增强在医学图像MRI与CT的域自适应任务中对分割结果有一定改善.
本节分析损失函数中系数δ 、γ 、$\epsilon$取不同值时对性能的影响, 其中δ 表示TDGM中的图像重构损失lrecon的权重, γ 表示共有特征鉴别器损失ladv的权重, $\delta$表示TDGM中对比损失lcons的权重.取
$\begin{array}{l} \delta=0.2, 0.6, 1.2, 2.4, \\ \gamma=0.2, 0.6, 1.2, 2.4 \\ \epsilon=0.1, 0.5, 1.0, 2.0 \end{array}$
具体指标值结果如表8所示, 表中黑体数字表示最优值.
| 表8 参数灵敏度分析结果 Table 8 Results of parameter sensitivity analysis |
由表8可见, 在腹部器官MRI→ CT分割任务上, 平均Dice值最高为85.3%, 最低为83.8%, 相差1.5%.平均ASD值最高为1.6, 最低为1.3, 相差0.3.当δ =0.2, γ =0.6, $\epsilon$=0.5时, Dice值最高.在心脏结构CT→ MRI分割任务中, 平均Dice值最高为79.9%, 最低为78.4%, 相差1.5%.平均ASD值最高为4.2, 最低为3.1, 相差1.1.当δ =0.2, γ =0.6, $\epsilon$=1.0时, 效果最优.上述数值说明TreUCMIS训练结果的稳定性较强.
本文提出基于目标域增强表示的医学图像无监督跨域分割方法(TreUCMIS).首先通过共有特征学习模块(SFLM)提取公有特征.针对特征对齐方法对齐后的共有特征偏向源域的问题, 设计共有特征增强模块(SFEM), 在输出空间使用一致性正则化方法, 增大共有特征空间, 缓解共有特征的偏向性问题.针对目前UDA忽略目标域私有特征的问题, 通过图像重构与一致性正则化方法, 使模型学会使用目标域图像的完整特征进行结果预测.在公开的腹部器官数据集和心脏结构数据集上的实验验证TreUCMIS的优越性.TDGM较依赖基于共有特征的预测结果的准确性, 如果基于共有特征的预测结果较差, 可能导致模型难以学习利用目标域图像的完整特征进行预测.今后将考虑研究更合适的目标域泛化方法和特征融合方式, 更好地利用目标域图像的共有特征和私有特征.此外, 还考虑将方法扩展到3D空间, 利用相邻切片的信息进行更准确的预测.
本文责任编委 杨健
Recommended by Associate Editor YANG Jian
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|
| [32] |
|
| [33] |
|
| [34] |
|
| [35] |
|
| [36] |
|
| [37] |
|